基于 PacBio SMRT 单分子测序技术的超长读长优势,解决了短读长的产品只能针对局部可变区进行分析的技术难题在合理成本的基础上,实现真正精确的菌群分类鉴定。
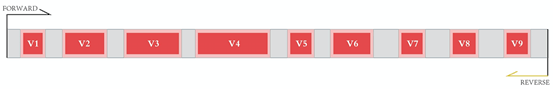
原核生物的16S rRNA基因全长(约1,500bp)分为9个可变区,基于PacBio全长的16S测序能够覆盖全部可变区,实现精确到“种”的分类鉴定
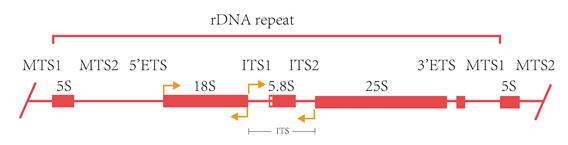
对于真核微生物来说,可以进行18S和ITS两个区域的全长测序来进行微生物群体分析
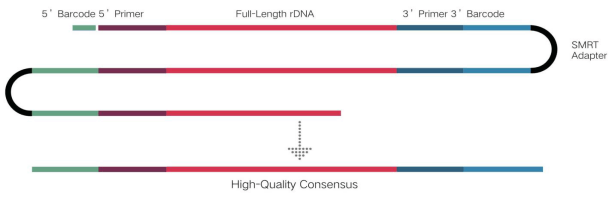
基于CCS Reads的原始错误矫正,能够得到精确度99%以上的全长rDNA,这是精确分类鉴定的保证
实验策略
| 建库测序 | 参考指标 | 交付周期 |
|---|---|---|
|
对环境样本基因组DNA进行 16S/18S/ITS/功能基因全长扩增, 构建文库PacBio Sequel平台测序 |
获取全长16S序列 每个样本约5000-20000个CCS reads |
建库测序:20个工作日 标准分析:5个工作日 高级分析:10个工作日 个性化分析:根据实际的信息分析内容而定 |
| 标准分析 | 原始数据处理 | |
| 基础分析 |
1. OTU聚类分析 2. OTU分类鉴定 3. alpha多样性指数分析 4. 测序量饱和度分析 5. 取样量饱和度分析 6. 秩和曲线 |
|
| 群落组成分析 |
1. 韦恩图 2. 群落组成分析 3. Megan分类学组成树 |
|
| 差异比较分析 |
1. 样本间差异计算:beta多样性分析 2. 多样本层次聚类树 3. 主成分分析 4. 主坐标分析 5. 非度量多维尺度分析 6. 约束性排序分析 7. 相似性分析 8. 方差分析 9.组间差异判别分析 |
|
| 高级分析 | 系统进化树 | |
| 基于进化距离的差异分析 | ||
| 网络关联分析 | ||